Par Carlos Lopez-Vaamonde – Antoine Guiguet – Rodolphe Rougerie
Des projections alarmantes de la perte de biodiversité indiquent que nous sommes entrés dans la sixième extinction de masse. Pendant ce temps, seule une petite fraction des espèces sur Terre a été officiellement décrite et a reçu un nom scientifique. Parmi ces organismes, les insectes étant à la fois fortement touchés par ce déclin mondial et contenant un grand nombre de espèces inconnues pour la science représentent un défi majeur pour la taxonomie, dont les méthodes classiques de description d’espèces sont souvent très longues.
Pour contourner ce verrou taxonomique, plusieurs techniques ont été développées dont les codes-barres ADN (ou DNA barcoding – lien). Le principe est d’utiliser un court fragment standard du génome et de le comparer à d’autres séquences identifiées au sein de librairies de références. Ainsi, ces bases de données de codes-barres ADN représentent un système global d’identification moléculaire. La plus développée aujourd’hui est BOLD (Barcode of Life Data Systems www.boldsystems.org) ; elle contient déjà plus de 8 millions de séquences représentant un demi-million d’espèces.
Cependant, ce déficit de connaissance taxonomique, appelé aussi déficit Linnéen, ne touche pas tous les groupes d’insectes de façon égale. Si la faune européenne de papillons est une des mieux connues au monde, le groupe des microlépidoptères qui comporte des papillons ne mesurant que quelques millimètres a suscité un plus faible intérêt de la part des taxonomistes du fait de leur difficulté d’étude. Un consortium de 19 chercheurs de 12 pays différents dirigé par Carlos Lopez-Vaamonde de INRAE Orléans a évalué l’utilité de cette approche « barcoding » pour l’identification d’espèces de la faune européenne de microlépidoptères de la famille des Gracillariidae (Lopez-Vaamonde et al. 2021) (figure 1).

Cette famille comporte 263 espèces décrites pour l’Europe, en faisant l’une des principales familles de microlépidoptères dans cette région. Appelées « mineuses de feuilles », les larves de Gracillariidae se nourrissent à dans l’épaisseur des feuilles en y creusant des mines, certaines espèces constituant des ravageurs importants tels que la mineuse du marronnier (Cameraria ohridella) et la mineuse du tilleul (Phyllonorycter issikii).
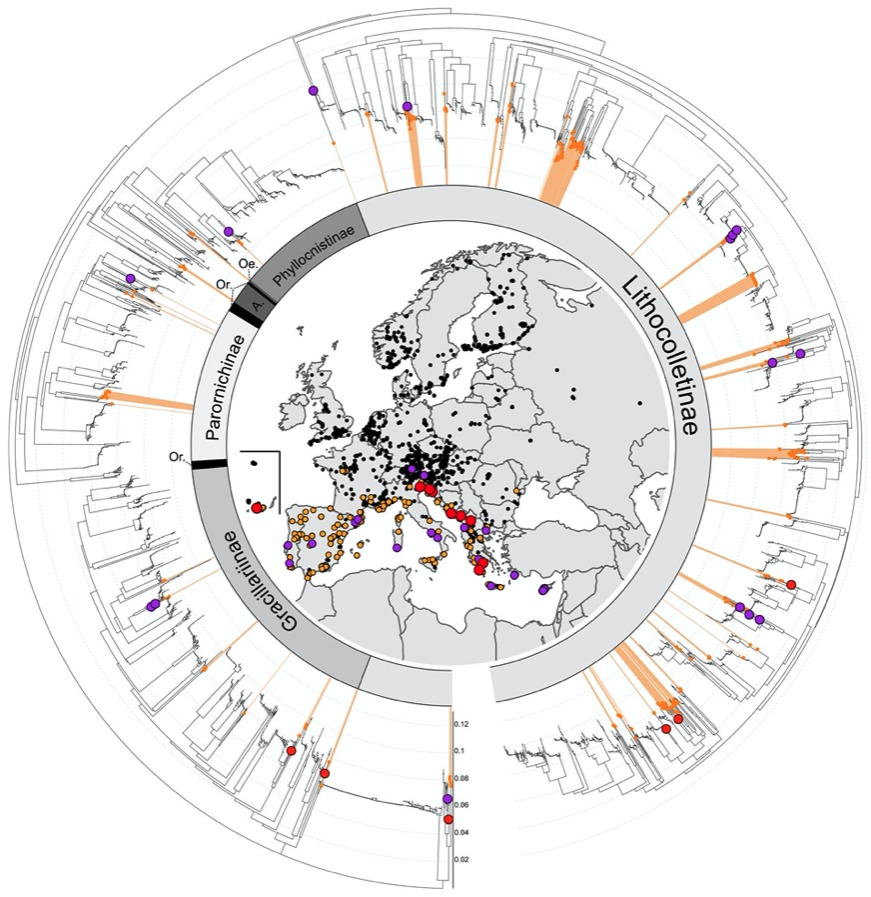
L’étude a permis de recueillir 6 791 séquences codes-barres du gene COI représentant 242 des 263 espèces résidentes, soit 92% de l’ensemble des Gracillariidae européens connus. Les données génétiques ont permis de découvrir six espèces nouvelles pour la science desormais en cours de description, ainsi que 15 autres espèces probablement nouvelles d’après leurs différences génétiques et écologiques et pour lesquelles il est nécessaire d’approfondir nos connaissances par l’étude d’un plus grand nombre de spécimens (figures 2 et 3).
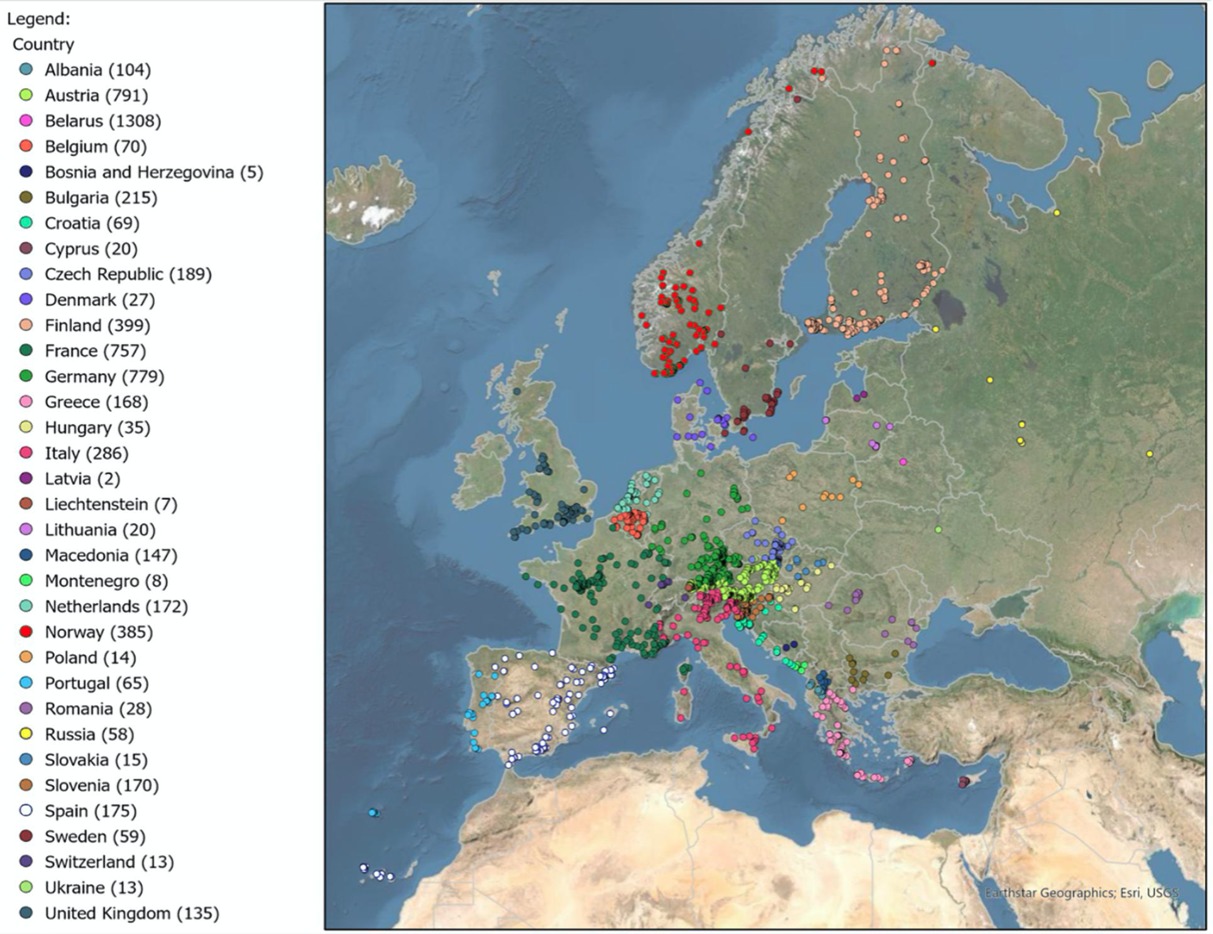
L’analyse des codes-barres ADN a aussi montré la présence potentielle d’espèces cryptiques (sans différentiation morphologique, mais avec divergence génétique) au sein de 26 espèces. Parmi les 242 espèces échantillonnées, il y a sept espèces non natives provenant de l’Amérique du Nord et l’Asie ainsi que 11 espèces européennes considérées comme ravageuses. Cette étude ouvre ainsi la voie à l’utilisation de cet outil génétique d’identification pour la biosurveillance de ces micro-papillons et de leurs larves.
Carlos Lopez-Vaamonde Carlos est entomologiste à l’Unité de Recherche de Zoologie Forestière (URZF – INRAE – Val-de-Loire) où il étudie l’écologie évolutive des insectes forestiers. Ses recherches se focalisent principalement sur la caractérisation des espèces et sur l’origine et l’évolution de la biodiversité forestière. Ces travaux utilisent une approche intégrative associant des données écologiques, biogéographiques, morphologiques et moléculaires. Il développe actuellement des protocoles de suivi des insectes forestiers à l’aide d’outils de métabarcording pour mieux comprendre comment la biodiversité forestière s’adapte aux changements globaux. |
Bibliographie
- Lopez-Vaamonde, C., Kirichenko, N., Cama, A., Doorenweerd, C., Godfray, C., Guiguet, A., Gomboc, S., Huemer, P., Landry, JF, Lastuvka, A., Lastuvka, Z., Kyungmin L., Lees, D., Mutanen, M., van Nieukerken E., Segerer, A., Triberti, P., Wieser, C. & Rougerie, R. (2021) : Evaluating DNA barcoding for species identification and discovery in European gracillariid moths. Frontiers in Ecology and Evolution9:626752 (lien)